《科學》:諾獎得主解讀 CRISPR 的十年
摘要:隨著生物學走入“分子時代”,基因編輯工具成為有力的科研武器,尤其是簇狀規則間隔短回文重復序列(CRISPR)基因編輯技術的出現,為基礎和應用生物學研究帶來了巨大的變革。
使用 CRISPR,我們能夠識別并快速地改變目標基因、診斷疾病、預測個體的疾病易感性,應用 CRISPR 的農業產物已經走進現實,CRISPR 邁出進入臨床醫學的第一步已然不遠……曾幾何時,人類基因組測序需 5 年才能完成;如今,24 小時內獲取完整序列已經變得可行。
CRISPR 是一門年輕的技術,它何以具有如此之大的能量?
近期,因開發 CRISPR 基因編輯技術于2020年獲得諾貝爾化學獎的 Jennifer Doudna 在《科學》雜志撰文[1],回顧了過去十年中 CRISPR 基因編輯的起源和應用、當前成果、局限性,并討論了未來的發展方向。就讓我們跟隨這篇文章一起來看看 CRISPR 的騰飛之旅。

圖1 CRISPR 的十年
改變生物學的技術
1987年,一篇論文首次將 CRISPR 帶入人類的視野[2]。CRISPR 是在微生物基因組中發現的一組規律重復的基因序列,它常與編碼 CRIPSR 相關蛋白(Cas)的基因一同出現。有趣的是,這些短重復序列竟然與病毒中的部分序列匹配[3]。
原來,CRISPR 系統其實是微生物的一種自我防御系統,通過 CRISPR 轉錄產物識別、Cas 蛋白切割來破壞病毒的 DNA 或 RNA,以此防御病毒入侵[4]。而這一運作系統的簡單和精巧使其具有了成為基因編輯工具的可能。
目前使用最廣泛的是被認為是第三代基因編輯技術的 CRISPR-Cas9 系統,由攜帶的引導 RNA(guide RNA)來識別序列,Cas9 蛋白(一種 RNA 指導的 DNA 內切核酸酶)切割 DNA,因此可以廣泛地靶向不同的序列。通過改變 Cas9 蛋白活性位點的氨基酸,也可以僅切斷 DNA 單鏈或者僅結合不切割,由此實現轉錄抑制、激活、沉默、上調等多種基因調控目的[5]。對 Cas 蛋白的進一步改造,也提供了單堿基編輯、染色質修飾、序列插入、靶向 RNA 等多樣的應用方向[6]。
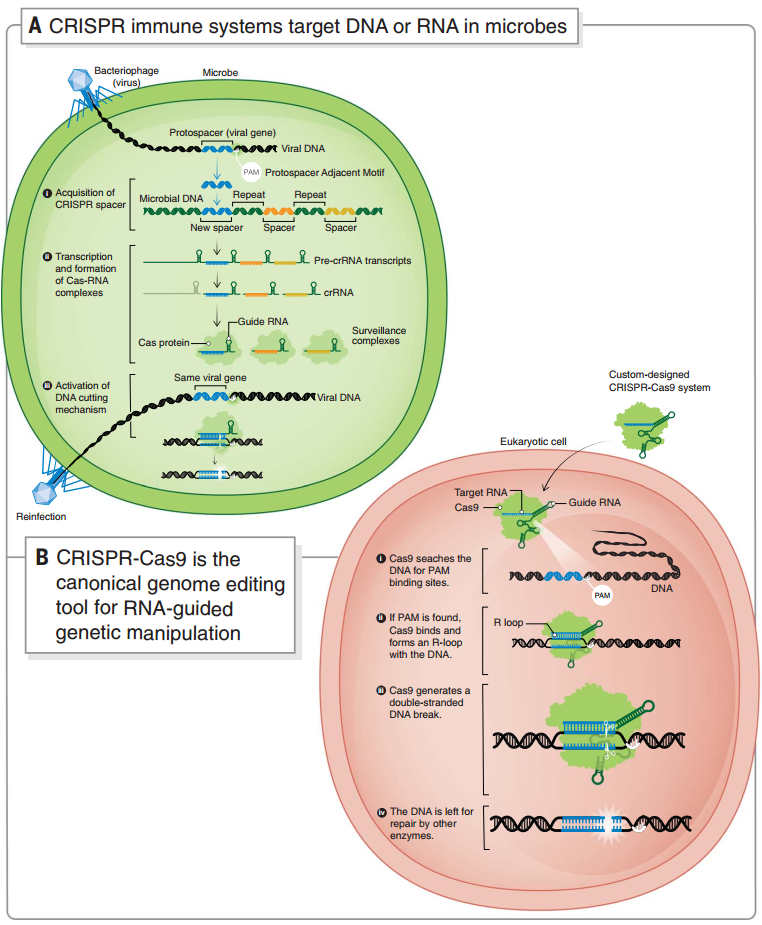
圖2 微生物中的 CRISPR 自我防御系統和 CRISPR-Cas9 基因編輯系統
CRISPR 技術的出現為多種疾病治療提供了基礎,包括鐮狀細胞病(SCD)、乙型地中海貧血、轉甲狀腺素蛋白(TTR)淀粉樣變性、先天性眼病等遺傳疾病,乃至癌癥、艾滋病毒感染等常見病;CRISPR 技術已經在農作物和家畜育種方面取得成果,比如更耐熱應激的奶牛(slick-coat cattle)及更有營養的番茄;而在基礎研究上,CRISPR 作為有力的科研工具,加速了分子和細胞生物學領域的研究,在數千篇論文中起到了作用。
接下來,就讓我們來具體了解一下 CRISPR 這個優秀的科研工具箱。
“萬能”工具箱
CRISPR 結構精巧靈活、可塑性極強,不同 CRISPR 體系具有多種功用,接下來我們選取其中使用較為廣泛的三類進行介紹。
①基因敲除
基因敲除是 CRISPR 最主要的應用之一,真核細胞中普遍使用 CRISPR-Cas9 系統,由工程單導 RNA(sgRNA)將 Cas9 核酸酶引導到目標位點,制造 DNA 雙鏈斷裂(DSB),由非同源末端連接(NHEJ)和同源定向修復(HDR)等內源性修復途徑修復。
這種技術已經廣泛應用于基因敲除動物模型的創建,由于繞過了傳統手段的復雜胚胎干細胞篩選過程,培育時間大大縮短,培育轉基因小鼠的時長從 1 年縮短到了 4 周[7]。同時,大多數哺乳動物并沒有成熟的胚胎干細胞系,通過 CRISPR-Cas9 也能夠促進新物種的動物模型出現。
在生殖細胞編輯之外,CRISPR-Cas9 也可以用于體細胞編輯,這避免了部分基因全身敲除的致命影響,在特定場景下也更有利于模擬癌癥的演變和進展以及確定癌癥治療的新策略[8]。
利用 CRISPR,研究者已經制造了多種疾病的動物模型,包括酪氨酸血癥、杜氏肌營養不良、癌癥、骨質疏松、亨廷頓舞蹈癥、肌萎縮型側索硬化癥、阿爾茨海默病和艾滋病等[9]。
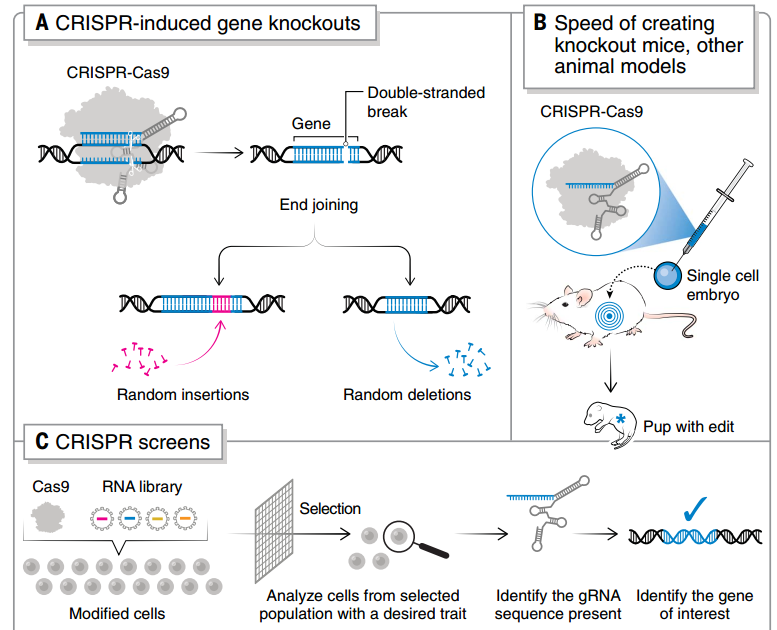
圖3 CRISPR 敲除和 CRISPR 篩選的流程
② CRISPR 篩選
當通過 CRISPR 并行執行針對多個基因的遺傳改變,就是 CRISPR 篩選。由于靈活高效,CRISPR 可以方便地引入基因擾動,可以深入研究單個基因擾動如何影響目標細胞的功能,又或是在混合篩選中對數千個擾動進行高通量測序[10]。當 gRNA 文庫達到基因組規模,研究者可以輕易使用 CRISPR 來擾動人類基因組中的每一個基因[11]。
在 CRISPR 敲除(CRISPR KO)篩選之外,CRISPR 干擾(CRISPRi)篩選和 CRISPR 激活(CRISPRa)篩選也是常用的可逆的控制基因表達的篩選方法[12];飽和基因組編輯(saturation genome editing)能夠產生所有可能的單核苷酸多態性(SNPs)用于功能篩選[13];后文會介紹的單堿基編輯技術能夠以更高效率引入點突變,或許會成為 CRISPR 篩選的新助力。
CRISPR 篩選已經在類器官、動物、植物等復雜模型中實際應用,它目前是分析癌基因功能的常用方法,還可以用于識別各種癌癥驅動因素和調節因素[10]。
結合其他技術,CRISPR 篩選同樣可用于細胞表型篩選、腫瘤耐藥性篩選等等領域,比如 Procode、Perturb-ATAC、Perturb-seq 和 ECCITE-seq 等多組學篩選技術。
③單堿基編輯技術
單堿基編輯技術提供了一種在不切斷 DNA 雙鏈的情況下進行基因編輯的新可能,這一方面可以產生精確的點突變,一方面無需復制模板且限制了多余的副產物。
單堿基編輯系統通常由不具有催化 DSB 活性的 dCas9、dCas12a、dCas13b 等 Cas 蛋白和堿基脫氨酶融合組成,sgRNA 將融合酶引導到目標位點,脫氨酶進行化學修飾,通過細胞修復機制處理堿基不匹配。在過去的幾年里,DNA 和 RNA 單堿基編輯已經實現了 C>T、A>G、C>G、A>I和C>U 轉換突變。
點突變是人類致病遺傳變異的主要類別,單堿基編輯技術為治療人類遺傳疾病鋪平了道路。單堿基編輯已經在多種小鼠模型中表現出了糾正功能喪失突變的良好效果,例如體內單堿基編輯糾正小鼠 Hutchinson-Gilford 早衰綜合征[14]。使用單堿基編輯技術治療家族性高膽固醇血癥的早期臨床試驗已經開始,近期將開展的還有針對鐮狀細胞病的臨床研究[15]。
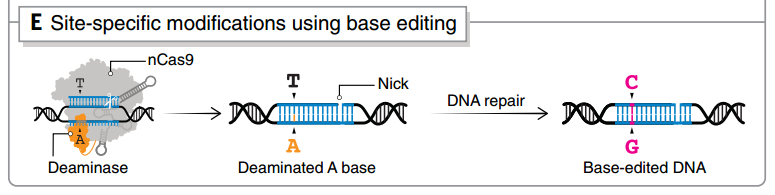
圖4 單堿基編輯技術之一base-editing的原理
CRISPR 進化的方向
隨著 CRISPR 基因編輯技術進入下一個十年,還有一些關鍵的問題需要解決,其中一個就是編輯的精度。這既指編輯目標位點的特異性,也指編輯結果的準確性。
為減少意外結合和切割導致的脫靶效應,研究者利用合理的設計開發了高保真Cas變體,例如SpCas9-HF1、evoCas9、HiFiCas9和 Cas9_R63A/Q768A 等變體,亦或是設計了優化的導向方法,例如 E-Crisp、CasOFFinder、sgDesigner。近期,也有研究開發了機器學習工具來預測基因編輯結果,但目前尚未得到體內研究的驗證[16]。
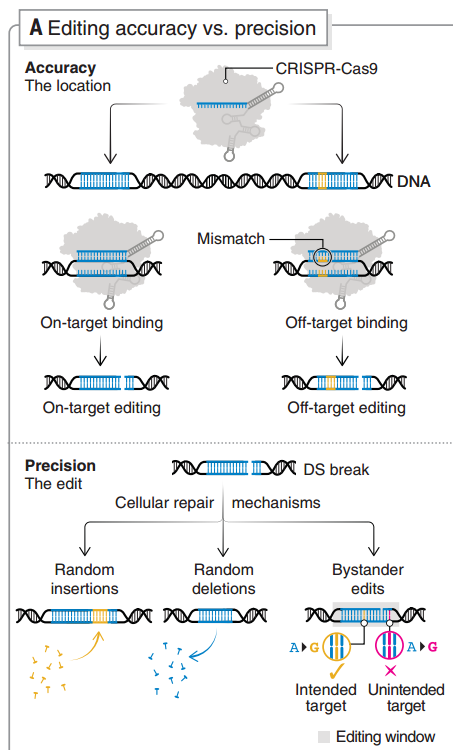
圖5 基因編輯特異性和準確性
傳統編輯中,DSB 后涉及 NHEJ 或 HDR 通路的修復(前者容易出錯,可能導致突變,后者雖更精確,但發生率非常低),這會導致一系列插入缺失突變(indels)。避免這個問題的其中一個思路就是提高 HDR 效率和/或抑制NHEJ,例如使用單鏈寡脫氧核苷酸模板[17]、通過控制細胞周期階段來促進 HDR [18]、使用位點特異性 Cas9 寡核苷酸偶聯物將 DNA 模板募集到目標位點[19]。但在這些方法之下仍存在 DSB 相關的大量基因位點缺失和染色體重排風險,可能導致基因組不穩定。
避免產生DSB則是另一個思路,堿基編輯(base editing,BE)和引導編輯(prime editing,PE)是目前的主要方法。不過 BE 也存在旁觀者編輯的問題,當編輯窗口中存在多個目標堿基時,BE 的編輯精度會大大降低[20]。Cas 蛋白識別特定原間隔序列鄰近基序(PAM),受此限制,縮小編輯窗口也存在一定困難,這還需要結合多種輔助策略來改善。PE 同樣不涉及 DSB,且能夠實現 DNA 序列的插入,但目前 PE 的編輯效率還比較低[21]。
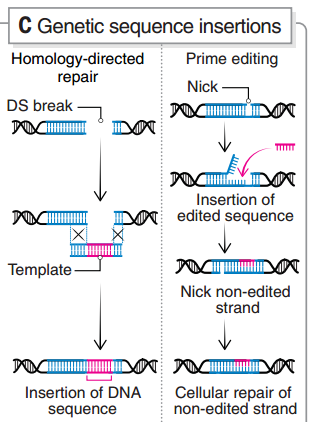
圖6 引導編輯的原理
限制 CRIPSR 應用的另一個環節是 CRISPR 的遞送。目前在哺乳動物系統中存在多種遞送方法,可以大致分為物理遞送、病毒遞送和基于合成材料的遞送。常用的物理方法包括微注射和電穿孔,傳遞效率高且劑量可控,但這局限于體外遞送;病毒遞送常用腺相關病毒(AAV)、腺病毒(AdVs)和慢病毒等載體,其中 AAV 在臨床研究中應用較多,但病毒載體存在載量、免疫原性、制造成本等多種潛在問題;脂質納米顆粒(LNPs)、陽離子聚合物、多肽和納米金(gold nanoparticles,GNPs)等合成材料載體通常較為安全可控,LNPs 是首次成功用于治療TTR淀粉樣變性的遞送載體[22]。
體內環境對 CRISPR 遞送具有多種阻礙,載體需避免降解、調理素作用、吞噬導致從血管滲出,有效穿過狹窄間隙,并在內吞時釋放內含物。CRISPR 治療人類疾病能夠發展到何種程度,很大程度上將取決于當前遞送方法的改進、新遞送方法的創建和設計更緊湊的 CRISPR 編輯系統。
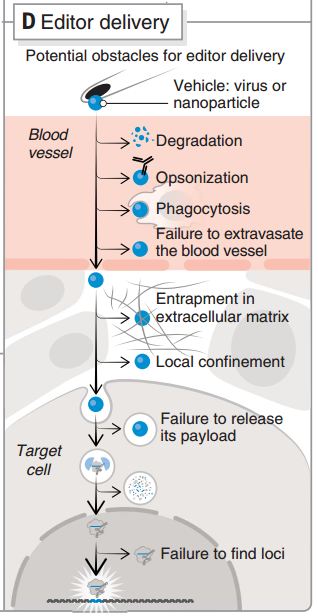
圖7 CRISPR 遞送過程
結語
可編程基因編輯技術的出現為細胞和基因治療的應用鋪平了道路。已有大量應用 CRISPR 的臨床試驗正在進行,其中關于 SCD 的至少已有 8 項正在進行或即將開始,預計 2023 年內 FDA 會啟動至少一項療法的批準[23]。隨著臨床應用的擴大,CRISPR 也有可能成為預防神經退行性疾病和心血管疾病的一種手段[24]。
CIRSPR 也對農業和畜牧業產生了深遠的影響,CRISPR 編輯的食品已經進入市場,包括營養價值更高的番茄和兩種已獲批在日本銷售的魚類。CRISPR 精準編輯的特點非常適合定向育種,可以同時敲除和激活不同的基因,例如給小麥引入抗病性的同時增加小麥的產量[25]。
CRISPR-Cas9 和 CRISPR-Cas12a 是目前應用最廣的編輯系統,已經在全世界各地的實驗室得到了廣泛的應用。這加速了基礎研究的進展,也倒推了 CRISPR 工具箱的擴展。結合機器學習、活細胞成像、DNA 測序技術, CRISPR 還能做到更多。
下一個十年,CIRSPR 還將繼續改變世界。
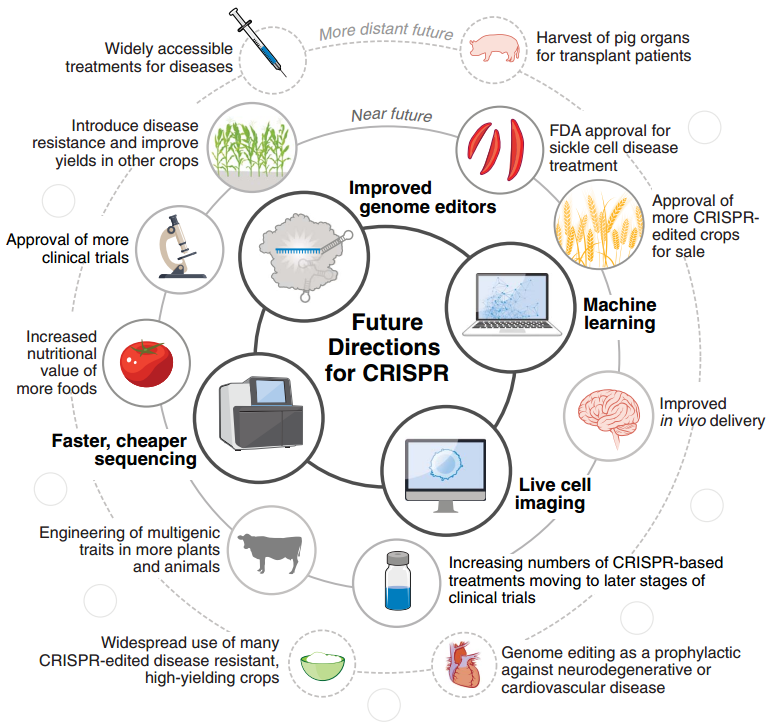
圖8 未來的 CRISPR
參考資料:
[1] Wang J Y, Doudna J A. CRISPR technology: A decade of genome editing is only the beginning[J]. Science, 2023, 379(6629): eadd8643.
[2] Y. Ishino, H. Shinagawa, K. Makino, M. Amemura, A. Nakata,Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J. Bacteriol. 169,5429–5433 (1987). doi: 10.1128/jb.169.12.5429-5433.1987;pmid: 3316184
[3] F. J. M. Mojica, C. Díez-Villaseñor, J. García-Martínez, E. Soria,Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J. Mol. Evol.60, 174–182 (2005). doi: 10.1007/s00239-004-0046-3;pmid: 15791728
[4] S. J. J. Brouns et al., Small CRISPR RNAs guide antiviral defense in prokaryotes. Science 321, 960–964 (2008).doi: 10.1126/science.1159689; pmid: 18703739
[5] L. S. Qi et al., Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression.Cell 152, 1173–1183 (2013). doi: 10.1016/j.cell.2013.02.022;pmid: 23452860
[6] J. Y. Wang, P. Pausch, J. A. Doudna, Structural biology of CRISPR-Cas immunity and genome editing enzymes.Nat. Rev. Microbiol. 20, 641–656 (2022). doi: 10.1038/s41579-022-00739-4; pmid: 35562427
[7] H. Yang, H. Wang, R. Jaenisch, Generating genetically modified mice using CRISPR/Cas-mediated genome engineering. Nat. Protoc. 9, 1956–1968 (2014). doi: 10.1038/nprot.2014.134; pmid: 25058643
[8] H. Mou, Z. Kennedy, D. G. Anderson, H. Yin, W. Xue, Precision cancer mouse models through genome editing with CRISPRCas9. Genome Med. 7, 53 (2015). doi: 10.1186/s13073-015-0178-7; pmid: 26060510
[9] H. Lee, D. E. Yoon, K. Kim, Genome editing methods in animal models. Anim. Cells Syst. 24, 8–16 (2020). doi: 10.1080/19768354.2020.1726462; pmid: 32158611
[10] A. Katti, B. J. Diaz, C. M. Caragine, N. E. Sanjana, L. E. Dow,CRISPR in cancer biology and therapy. Nat. Rev. Cancer 22,259–279 (2022). doi: 10.1038/s41568-022-00441-w;pmid: 35194172
[11] N. E. Sanjana, O. Shalem, F. Zhang, Improved vectors and genome-wide libraries for CRISPR screening. Nat.Methods 11, 783–784 (2014). doi: 10.1038/nmeth.3047;pmid: 25075903
[12] L. Przybyla, L. A. Gilbert, A new era in functional genomics screens. Nat. Rev. Genet. 23, 89–103 (2022). doi: 10.1038/s41576-021-00409-w; pmid: 34545248
[13] G. M. Findlay et al., Accurate classification of BRCA1 variants with saturation genome editing. Nature 562, 217–222 (2018).doi: 10.1038/s41586-018-0461-z; pmid: 30209399
[14] L. W. Koblan et al., In vivo base editing rescues Hutchinson-Gilford progeria syndrome in mice. Nature 589, 608–614 (2021). doi: 10.1038/s41586-020-03086-7;pmid: 33408413
[15] K. Kingwell, Base editors hit the clinic. Nat. Rev. Drug Discov.21, 545–547 (2022). doi: 10.1038/d41573-022-00124-z;pmid: 35831515
[16] W. Chen et al., Massively parallel profiling and predictive modeling of the outcomes of CRISPR/Cas9-mediated doublestrand break repair. Nucleic Acids Res. 47, 7989–8003(2019). doi: 10.1093/nar/gkz487; pmid: 31165867
[17] C. D. Richardson, G. J. Ray, M. A. DeWitt, G. L. Curie,J. E. Corn, Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nat. Biotechnol. 34, 339–344(2016). doi: 10.1038/nbt.3481; pmid: 26789497
[18] A. Lomova et al., Improving gene editing outcomes in human hematopoietic stem and progenitor cells by temporal control of DNA repair. Stem Cells 37, 284–294 (2019).doi: 10.1002/stem.2935; pmid: 30372555
[19] X. Ling et al., Improving the efficiency of precise genome editing with site-specific Cas9-oligonucleotide conjugates.Sci. Adv. 6, eaaz0051 (2020). doi: 10.1126/sciadv.aaz0051;pmid: 32494588
[20] M. Arbab et al., Determinants of base editing outcomes from target library analysis and machine learning. Cell 182, 463–480.e30 (2020). doi: 10.1016/j.cell.2020.05.037; pmid: 32533916
[21]M. M. Hassan, G. Yuan, J.-G. Chen, G. A. Tuskan, X. Yang,Prime editing technology and its prospects for future applications in plant biology research. BioDesign Research 2020, 1–14 (2020). doi: 10.34133/2020/9350905
[22] J. D. Gillmore et al., CRISPR-Cas9 In vivo gene editing for transthyretin amyloidosis. N. Engl. J. Med. 385, 493–502(2021). doi: 10.1056/NEJMoa2107454; pmid: 34215024
[23] F. D. Urnov, The Cas9 hammer-and sickle: A challenge for genome editors. CRISPR J. 4, 6–13 (2021). doi: 10.1089/crispr.2021.29120.fur; pmid: 33616446
[24] K. Musunuru et al., In vivo CRISPR base editing of PCSK9 durably lowers cholesterol in primates. Nature 593,429–434 (2021). doi: 10.1038/s41586-021-03534-y;pmid: 34012082
[25] S. Li et al., Genome-edited powdery mildew resistance in wheat without growth penalties. Nature 602, 455–460(2022). doi: 10.1038/s41586-022-04395-9;pmid: 35140403
[2] Y. Ishino, H. Shinagawa, K. Makino, M. Amemura, A. Nakata,Nucleotide sequence of the iap gene, responsible for alkaline phosphatase isozyme conversion in Escherichia coli, and identification of the gene product. J. Bacteriol. 169,5429–5433 (1987). doi: 10.1128/jb.169.12.5429-5433.1987;pmid: 3316184
[3] F. J. M. Mojica, C. Díez-Villaseñor, J. García-Martínez, E. Soria,Intervening sequences of regularly spaced prokaryotic repeats derive from foreign genetic elements. J. Mol. Evol.60, 174–182 (2005). doi: 10.1007/s00239-004-0046-3;pmid: 15791728
[4] S. J. J. Brouns et al., Small CRISPR RNAs guide antiviral defense in prokaryotes. Science 321, 960–964 (2008).doi: 10.1126/science.1159689; pmid: 18703739
[5] L. S. Qi et al., Repurposing CRISPR as an RNA-guided platform for sequence-specific control of gene expression.Cell 152, 1173–1183 (2013). doi: 10.1016/j.cell.2013.02.022;pmid: 23452860
[6] J. Y. Wang, P. Pausch, J. A. Doudna, Structural biology of CRISPR-Cas immunity and genome editing enzymes.Nat. Rev. Microbiol. 20, 641–656 (2022). doi: 10.1038/s41579-022-00739-4; pmid: 35562427
[7] H. Yang, H. Wang, R. Jaenisch, Generating genetically modified mice using CRISPR/Cas-mediated genome engineering. Nat. Protoc. 9, 1956–1968 (2014). doi: 10.1038/nprot.2014.134; pmid: 25058643
[8] H. Mou, Z. Kennedy, D. G. Anderson, H. Yin, W. Xue, Precision cancer mouse models through genome editing with CRISPRCas9. Genome Med. 7, 53 (2015). doi: 10.1186/s13073-015-0178-7; pmid: 26060510
[9] H. Lee, D. E. Yoon, K. Kim, Genome editing methods in animal models. Anim. Cells Syst. 24, 8–16 (2020). doi: 10.1080/19768354.2020.1726462; pmid: 32158611
[10] A. Katti, B. J. Diaz, C. M. Caragine, N. E. Sanjana, L. E. Dow,CRISPR in cancer biology and therapy. Nat. Rev. Cancer 22,259–279 (2022). doi: 10.1038/s41568-022-00441-w;pmid: 35194172
[11] N. E. Sanjana, O. Shalem, F. Zhang, Improved vectors and genome-wide libraries for CRISPR screening. Nat.Methods 11, 783–784 (2014). doi: 10.1038/nmeth.3047;pmid: 25075903
[12] L. Przybyla, L. A. Gilbert, A new era in functional genomics screens. Nat. Rev. Genet. 23, 89–103 (2022). doi: 10.1038/s41576-021-00409-w; pmid: 34545248
[13] G. M. Findlay et al., Accurate classification of BRCA1 variants with saturation genome editing. Nature 562, 217–222 (2018).doi: 10.1038/s41586-018-0461-z; pmid: 30209399
[14] L. W. Koblan et al., In vivo base editing rescues Hutchinson-Gilford progeria syndrome in mice. Nature 589, 608–614 (2021). doi: 10.1038/s41586-020-03086-7;pmid: 33408413
[15] K. Kingwell, Base editors hit the clinic. Nat. Rev. Drug Discov.21, 545–547 (2022). doi: 10.1038/d41573-022-00124-z;pmid: 35831515
[16] W. Chen et al., Massively parallel profiling and predictive modeling of the outcomes of CRISPR/Cas9-mediated doublestrand break repair. Nucleic Acids Res. 47, 7989–8003(2019). doi: 10.1093/nar/gkz487; pmid: 31165867
[17] C. D. Richardson, G. J. Ray, M. A. DeWitt, G. L. Curie,J. E. Corn, Enhancing homology-directed genome editing by catalytically active and inactive CRISPR-Cas9 using asymmetric donor DNA. Nat. Biotechnol. 34, 339–344(2016). doi: 10.1038/nbt.3481; pmid: 26789497
[18] A. Lomova et al., Improving gene editing outcomes in human hematopoietic stem and progenitor cells by temporal control of DNA repair. Stem Cells 37, 284–294 (2019).doi: 10.1002/stem.2935; pmid: 30372555
[19] X. Ling et al., Improving the efficiency of precise genome editing with site-specific Cas9-oligonucleotide conjugates.Sci. Adv. 6, eaaz0051 (2020). doi: 10.1126/sciadv.aaz0051;pmid: 32494588
[20] M. Arbab et al., Determinants of base editing outcomes from target library analysis and machine learning. Cell 182, 463–480.e30 (2020). doi: 10.1016/j.cell.2020.05.037; pmid: 32533916
[21]M. M. Hassan, G. Yuan, J.-G. Chen, G. A. Tuskan, X. Yang,Prime editing technology and its prospects for future applications in plant biology research. BioDesign Research 2020, 1–14 (2020). doi: 10.34133/2020/9350905
[22] J. D. Gillmore et al., CRISPR-Cas9 In vivo gene editing for transthyretin amyloidosis. N. Engl. J. Med. 385, 493–502(2021). doi: 10.1056/NEJMoa2107454; pmid: 34215024
[23] F. D. Urnov, The Cas9 hammer-and sickle: A challenge for genome editors. CRISPR J. 4, 6–13 (2021). doi: 10.1089/crispr.2021.29120.fur; pmid: 33616446
[24] K. Musunuru et al., In vivo CRISPR base editing of PCSK9 durably lowers cholesterol in primates. Nature 593,429–434 (2021). doi: 10.1038/s41586-021-03534-y;pmid: 34012082
[25] S. Li et al., Genome-edited powdery mildew resistance in wheat without growth penalties. Nature 602, 455–460(2022). doi: 10.1038/s41586-022-04395-9;pmid: 35140403
 |
 |
 |
| 官網:m.baichuan365.com | 微信服務號:iseebio | 微博:seebiobiotech |
 |
 |
 |
| 商城:mall.seebio.cn | 微信訂閱號:seebiotech | 泉養堂:www.canmedo.com |
此文關鍵字:CRISPR基因編輯技術
相關資訊
- MIT神經學大牛關于阿爾茨海默病的超級顛覆研究
- 杭師大:miRNA調控水稻葉片衰老
- CTI:深度揭示COVID-19患者的免疫反應特征
- 8分鐘檢測A群鏈球菌感染 Alere分子診斷試劑獲FDA批準
- 皮膚素硫酸鹽低聚糖標準品
- 「西寶生物」專業提供美國NU-CHEK【脂肪酸標準品】
- 孩子最喜歡吃的彩虹糖可致DNA改變!傷害大腦、肝臟、腎臟……
- 環氧化磁珠
- 揭開白藜蘆醇的延年益壽之謎
- 干細胞“種”出血管化腐生肌 中山醫院保肢率達91.2%
新進產品
同類文章排行
- 《科學》:諾獎得主解讀 CRISPR 的十年
- 《Nature》甲流當季,大腦如何感知感染以及“布洛芬”沒效果的原因?
- 修改mRNA或可治療阿爾茨海默病
- Immunity:免疫系統的“馬拉松選手”
- 《PNAS》科學家解開細胞存活之謎
- 大腦越用越“廢”?Nature發現神經元DNA修復機制,或推動相關疾病研究進展
- 干細胞療法創新:新的水凝膠維持干細胞存活 可修復小鼠受傷腦組織
- 肺細胞如何保護自己免受RNA病毒感染?
- 《柳葉刀》:首次成功利用CAR-T細胞治療嚴重肌肉炎癥
- Science子刊:對過敏性哮喘的保護—先天淋巴樣細胞
資訊文章
您的瀏覽歷史